Низька варіабельність супутникової ДНК у природних популяціях Drosophila antonietae, залучених до різних еволюційних подій
Машинний переклад
Оригінальна стаття написана мовою EN (посилання для прочитання) .
Drosophila antonietae є кактусофільним видом, який зустрічається в мезофільному лісі басейну річки Парана–Парагвай та на дюнах південноатлантичного узбережжя Бразилії. Хоча генетична структура популяцій басейну річки Парана–Парагвай вже була встановлена, зв'язок між цими популяціями та популяціями на атлантичному узбережжі є суперечливим. У цьому дослідженні ми порівняли 33 повторювані одиниці супутникової ДНК pBuM-2, ізольовані від індивідів з 8 популяцій D. antonietae в цих географічних регіонах, включаючи деякі популяції, що знаходяться в зоні контакту з близькоспорідненою D. serido. Послідовності pBuM-2 показали низьку міжпопуляційну варіабельність. Цей результат був інтерпретований як наслідок як генетичного потоку між популяціями, так і нерівного кросинговеру, що сприяє гомогенізації тандемних масивів. Результати, представлені тут, разом з результатами попередніх досліджень, підкреслюють використання pBuM-2 для вирішення таксономічних конфліктів у межах кластеру видів D. buzzatii.
Вступ
Drosophila antonietae є членом групи видів D. buzzatii, монофілетичної групи, що складається з 7 споріднених видів, які природно ендемічні для неотропічного регіону, що зустрічаються в відкритих і сухих лісах на сході Південної Америки (Manfrin and Sene 2006). Ці види використовують некротичну тканину кактусів як місця для розмноження (Pereira et al. 1983); через це їх географічний ареал обмежений розподілом господарських кактусів. Таким чином, D. antonietae є привабливою моделлю для вивчення еволюційних процесів (Manfrin and Sene 2006).
Географічний розподіл D. antonietae можна охарактеризувати двома способами. У долинах, оточених басейном річки Парана–Парагвай, популяції знаходяться в мезофільних лісах, пов'язаних з кактусом Cereus hildemannianus (Tidon-Sklorz and Sene 2001; Mateus and Sene 2003). У своєму південно-східному розподілі D. antonietae зустрічається в дюнних регіонах Атлантичного узбережжя, використовуючи переважно C. hildemannianus, але також кактуси з роду Opuntia (Ruiz et al. 2000; Manfrin and Sene 2006) (Рисунок 1).
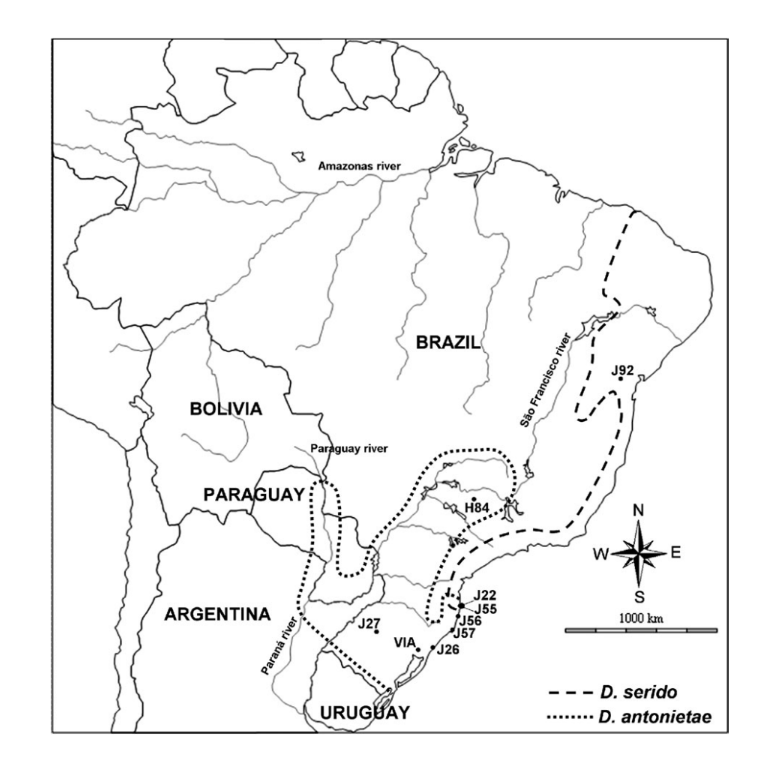
Попередні дослідження показали, що популяції D. antonietae з басейну річки Парана–Парагвай структуровані відповідно до моделі ізоляції за відстанню через позитивну кореляцію морфологічних і генетичних відстаней з географічними та екологічними відстанями (останні визначаються як відстань між 2 популяціями вздовж русла річки) (de Brito et al. 2002; Mateus and Sene 2007). На основі кількості мігрантів на покоління, Mateus and Sene (2007) запропонували помірний вплив як генетичного потоку, так і генетичного дрейфу в цих популяціях D. antonietae, з генетичним потоком, що перекриває генетичний дрейф, що сприяє генетичному градієнту.
Філогеографічний аналіз на основі мітохондріальної ДНК показав, що південно-східний ареал D. antonietae на атлантичному узбережжі Бразилії був колонізований внаслідок подій розширення популяцій D. antonietae з центральної депресії штату Ріо-Гранде-ду-Сул (de Brito et al. 2002; Manfrin and Sene 2006). Однак цей аналіз не зміг визначити, чи було це розширення супроводжене підтримкою генетичного потоку, з набуттям популяційної структури відповідно до моделі ізоляції за відстанню, чи супроводжене фрагментацією, що вказує на відсутність генетичного потоку між популяціями атлантичного узбережжя та тими, що з басейну річки Парана–Парагвай (de Brito et al. 2002). Дані про хромосомні інверсії можуть вказувати на певний ступінь фрагментації між популяціями з цих 2 різних середовищ, оскільки ті популяції з долин басейну річки Парана–Парагвай є поліморфними для інверсій 2y8, 2z8 та 5e, тоді як популяції з атлантичного узбережжя є поліморфними для 2z8 (Ruiz et al. 2000). Популяції D. antonietae на атлантичному узбережжі мають ще одну важливу характеристику: вони контактують з близькоспорідненим видом D. serido (D. buzzatii кластер) (Ruiz et al. 2000; Manfrin and Sene 2006). Ця зона перекриття є результатом розширення D. serido з півночі на південь атлантичного узбережжя та спільного географічного місця з
D. antonietae популяції (de Brito et al. 2002; Manfrin та Sene 2006) (Рисунок 1). Морфологічні ознаки та аналізи мітохондріальної ДНК свідчать про те, що гібридизація між
D. antonietae та D. serido відбулася в зоні контакту (C.K.B. Santos та колеги, неопубліковані дані).
Супутникові ДНК є високорепетитивними тандемно організованими послідовностями ДНК, які складають значну частину гетерохроматину еукаріотичних організмів (Charlesworth та ін. 1tt4; Palomeque та Lorite 2008). Супутники зазвичай розташовані поблизу центромерних регіонів і, менш часто, у теломерах (Ugarkovic та Plohl 2002; Kuhn та ін. 2008). Ці послідовності еволюціонують у злагоджений спосіб, тоді як основні молекулярні механізми, що беруть участь в їхній еволюції, це реплікація з ковзанням, нерівний кросинговер, конверсія генів та реплікація за допомогою обертового кола (Dover 1982; Charlesworth та ін. 1994).
Деякі з родин супутникової ДНК швидко еволюціонують, що дозволяє їх використовувати як молекулярні маркери в міжвидових порівняльних дослідженнях (Watabe et al. 1997; Picariello et al. 2002; Kuhn and Sene 2005). Однак використання цих послідовностей як молекулярних маркерів у популяційних дослідженнях було відносно недостатньо вивчене, незважаючи на те, що конвергентна еволюція була зафіксована в популяціях Cyprinodon variegates (Cyprinodontidae) (Elder and Turner 1994) та Acrossocheilus paradoxus (Cyprinidae) (Wu et al. 1999).
Три родини супутникової ДНК, спільні для видів кластера D. buzzatii, були формально описані: pBuM (Kuhn et al. 1999, 2008; Kuhn and Sene 2005), DBC-150 (Kuhn et al. 2007), та SSS139 (Franco et al. 2008). Родина pBuM складається з 2 підродин, pBuM-1 та pBuM-2 (Kuhn and Sene 2005; Kuhn et al. 2008). Підродина pBuM-2 складається з АТ-багатих мономерів довжиною 370 п.н., які мають якісні (фіксована нуклеотидна заміна) та кількісні (кількість копій) відмінності серед видів кластера D. buzzatii (Kuhn and Sene 2005; Kuhn et al. 2007, 2008).
У цій роботі ми демонструємо інтраспецифічну варіабельність супутникової ДНК pBuM-2 у D. antonietae, використовуючи популяції з 2 різних середовищ (басейн Парани–Парагваю та Атлантичне узбережжя), які мають різні еволюційні історії, пов'язані з підтриманням генетичного потоку в першому випадку та гібридизаційними подіями з близькоспорідненим видом D. serido у другому. Ми вирішили використовувати супутникову ДНК pBuM як молекулярний маркер, оскільки вона має 14 діагностичних нуклеотидних замін між D. antonietae та D. serido (Kuhn and Sene 2005). Таким чином, pBuM дозволяє таксономічну ідентифікацію зразків, ізольованих з контактної зони. Більше того, оскільки еволюційна динаміка послідовностей супутникової ДНК на інтраспецифічному рівні не є повністю зрозумілою, подальші емпіричні дані можуть сприяти кращому розумінню процесу, що лежить в основі еволюції супутникової ДНК (Ugarkovicand Plohl 2002; Palomeque and Lorite 2008).
Матеріали та методи
Зразки
Ми вивчали 8 бразильських популяцій D. antonietae (Рисунок 1). Три з цих популяцій розташовані в долинах басейну річки Парана–Парагвай: J2/ (Сантьяго, штат Ріо-Гранде-ду-Сул), Via (Віамау, штат Ріо-Гранде-ду-Сул) та H84 (Серрана, штат Сан-Паулу, номер доступу AY656609– AY656615; Кун і Сене 2005); а 5 розташовані на дюнах Атлантичного узбережжя, в межах контактної зони з
D. serido: J56 (Гаропаба, штат Санта-Катарина), J55 (пляж Армакан, Флорианополіс, штат Санта-Катарина), J22 (пляж Жоакіна, Флорианополіс, штат Санта-Катарина), J5/ (Лагуна, штат Санта-Катарина) та J26 (Осоріо, штат Ріо-Гранде-ду-Сул) (Рисунок 1). Послідовності D. antonietae були проаналізовані разом з послідовностями G pBuM-2 з D. serido (популяція Jf2—Мілагрес, штат Баїя, номер доступу AY656616–AY656621; Кун і Сене 2005), щоб ідентифікувати деякі події інтрогресії в популяціях з Атлантичного узбережжя.
Ізоляція ДНК супутника pBuM-2 та аналіз послідовності
Геномна ДНК від одного чоловічого екземпляра (диких мух) з кожної популяції була виділена за допомогою набору для очищення геномної ДНК Wizard (Promega). Геномна ДНК була піддана полімеразній ланцюговій реакції для ампліфікації копій родини pBuM-2 [праймери A2F (CGGAGTA-TTTTTCATTCGAC) та A2R (GGTATGCCATAAAG-AAGTCG)] відповідно до Kuhn et al. (2008). Отримані смуги приблизно 400 п.н. були елюйовані з гелю шляхом нічної інкубації в елюційній розчині (500 мМ NaAc; 1 мМ етилендіамінтетраоцтова кислота). Відновлені фрагменти були клоновані за допомогою набору для клонування з тупими кінцями pMOSBlue (RPN 5110; Amersham Pharmacia Biotech). Плазмідна ДНК була підготовлена відповідно до методології, описаної в Sambrook et al. (1989), а реакція шаблону ДНК для секвенування була підготовлена відповідно до інструкції набору BigDye Terminator Cycle Sequencing Ready Reaction Kit (PerkinElmer). Автоматичне секвенування ДНК було виконано на секвенаторі ABI Prism 377 (PerkinElmer).
Вирівнювання послідовностей було виконано в CLUSTALW 1.8 (Thompson et al. 1994) та відредаговано в BioEdit (Hall 1999). Ми використовували алгоритм та статистичну модель, запропоновану Betrán et al. (1997), для виявлення ділянок генного перетворення. Параметр Rm, який оцінює мінімальну кількість подій рекомбінації в зразку ДНК (Hudson and Kaplan 1985), використовувався для виявлення подій рекомбінації. Хоча цей параметр не вимірює специфічно нерівномірний кросовер, розумно припустити, що Rm матиме значення, вищі за нуль, якщо відбувся нерівномірний кросовер. У цьому сенсі ми використовуємо Rm як непряме свідчення нерівномірного кросоверу. Генетичні відстані були розраховані відповідно до p-відстані, яка є пропорцією нуклеотидних відмінностей між 2 послідовностями (тобто p 5 Np/N, де Np є відмінностями нуклеотидів, а N є загальною кількістю нуклеотидів). Також до даних були застосовані більш складні моделі відстані, з подібними результатами, як ті, що представлені p-відстанню (дані не показані). Відстань матриця була використана для створення фенограми за допомогою методу сусіднього приєднання (Saitou and Nei 1987). Розрахунки генетичної відстані та фенограми були виконані за допомогою програми MEGA 3.0 (Kumar et al. 2004).
Ми також дослідили патерни внутрішньовидової варіації в D. antonietae за допомогою аналізів молекулярної варіації (AMOVAs; Excoffier et al. 1992). Для AMOVA популяції D. antonietae були згруповані в 2 групи: Атлантичне узбережжя та басейн річки Парана–Парагвай. AMOVA була виконана за допомогою Arlequin 3.0 (Excoffier et al. 2005).
Результати та обговорення
Було отримано двадцять шість нових мономерів супутникової ДНК pBuM-2: 4 з місцевості J56 (номер доступу FJ935973– FJ93597G); 4 з місцевості Via (номер доступу FJt35t77–FJt35t80); 4 з місцевості J26 (номер доступу FJ935981–FJ935984); 3 з місцевості J5/ (номер доступу FJ935985–FJ935987); 5 з місцевості J2/ (номер доступу FJ935988–FJ935992); 2 з місцевості J55 (номер доступу FJ935993–FJ935994); та 4 з місцевості J22 (номер доступу FJ935995–FJ935998). Висока нуклеотидна схожість між послідовностями, отриманими в цій роботі, та тими, що були описані раніше (Kuhn and Sene 2005), підтверджує, що вони належать до родини супутникової ДНК pBuM-2.
Усі отримані послідовності мають первинну структуру, характерну для D. antonietae, і були згруповані в одній гілці фенограми разом з раніше описаними послідовностями pBuM-2 D. antonietae (Рисунок 2), навіть тими, що з популяцій у зоні контакту з D. serido, де було запропоновано гібридизацію між D. antonietae та D. serido з інтрогресією мітохондріальної ДНК (C.K.B Santos, F.M. Sene та M.H. Manfrin, неопубліковані дані). Ця ситуація може бути пов'язана з спостереженнями, що мітохондріальні гени схильні до інтрогресії легше, ніж ядерні послідовності (Dorado et al. 1992; Arnold 1993). Найбільш цитовані пояснення цього спостереження пов'язані з процесами цито-ядерного дисбалансу (Arnold 1993) та стерильністю гібридних самців (Aubert and Solignac 1990).
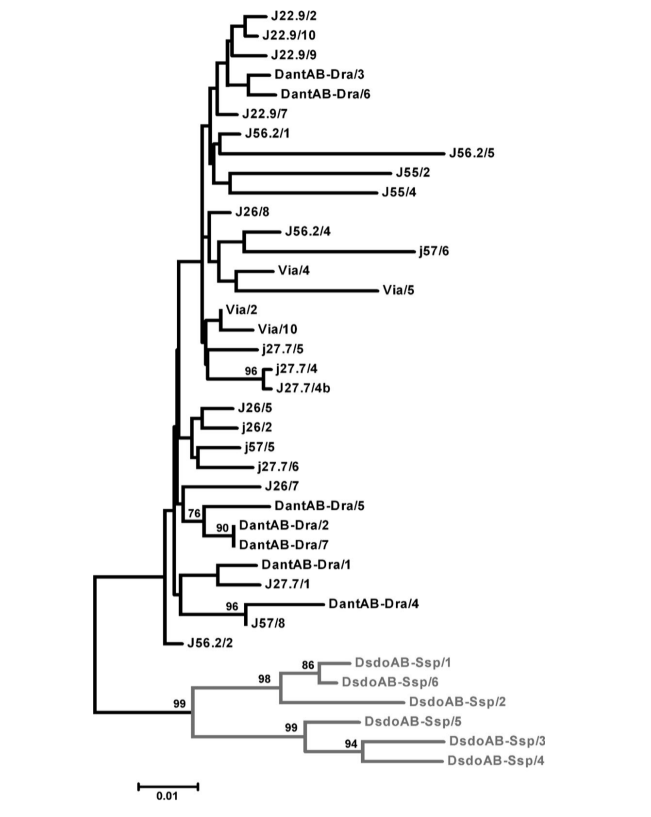
Існує високий рівень послідовнісної схожості серед послідовностей pBuM-2 D. antonietae (в середньому 95.7%), і жодні мутації не були виявлені як діагностичні для конкретної популяції. Міжпопуляційна варіабельність була дуже схожа на внутрішньопопуляційну варіабельність (Таблиця 1), і послідовності з однієї популяції не згрупувалися разом в конкретній гілці дендограмі (Рисунок 2). AMOVA, проведена з популяціями, розділеними за географічними групами (басейн річки Парана–Парагвай та Атлантичне узбережжя), показала, що 99.30% загальної генетичної різноманітності можна віднести до внутрішньогрупової варіабельності, тоді як лише 0.70% було віднесено до міжгрупової варіабельності (Таблиця 2). Розраховане Фst було низьким і не мало статистичної підтримки (Таблиця 2).
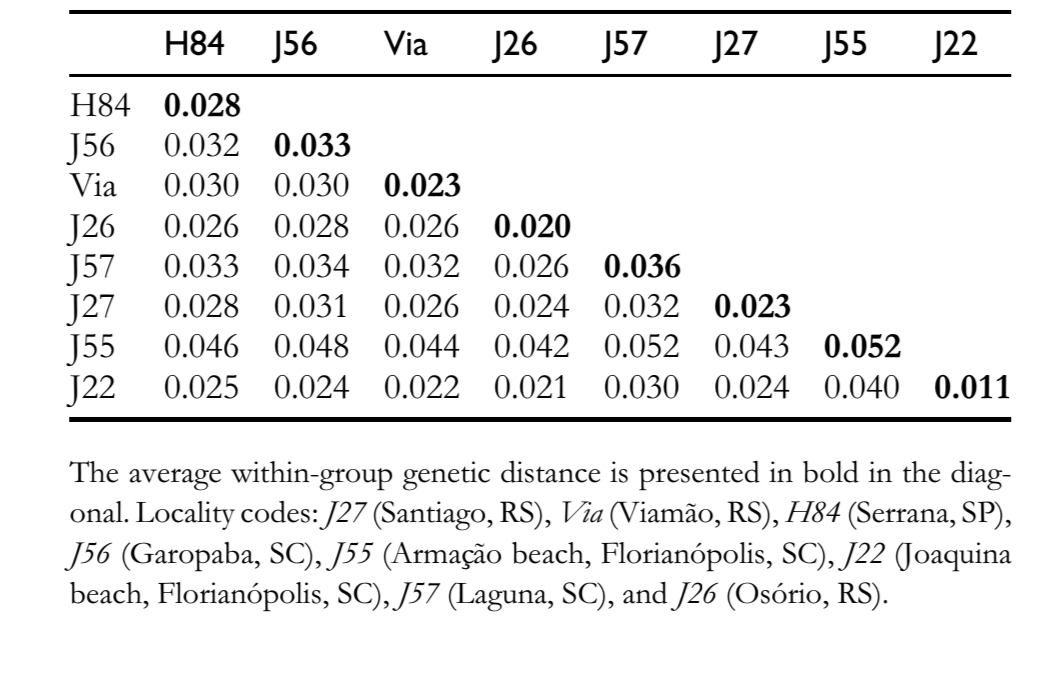

Популяції D. antonietae з басейну річки Парана–Парагвай структуровані відповідно до моделі ізоляції за відстанню (Монтейро та Сене 1995; Матейуш та Сене 2007). Спостережувані хромосомні інверсії свідчать про фрагментацію між популяціями з Атлантичного узбережжя та тими, що з басейну річки Парана–Парагвай (Руїз та ін. 2000), тоді як дані мітохондріальної ДНК не змогли визначити, чи була фрагментація або ізоляція за відстанню домінуючою структурою, що визначає взаємозв'язок цих 2 груп популяцій (де Бріто та ін. 2002). Висока нуклеотидна схожість pBuM-2 у D. antonietae (Рисунок 2 та Таблиці 1 і 2) узгоджується з підтримкою генетичного потоку між популяціями цього виду, навіть серед тих, що розташовані в басейні річки Парана́–Парагвай та на Атлантичному узбережжі. Наші результати свідчать про те, що ізоляція за відстанню повинна пояснити варіабельність мітохондріальної ДНК у D. antonietae; ця модель передбачає генетичний потік між популяціями, як це випливає з даних про супутню ДНК, представлених тут. У цьому контексті найнижчі поліморфізми хромосомних інверсій у популяціях з Атлантичного узбережжя D. antonietae, виявлені Руїзом та ін. (2000), можуть бути пояснені як наслідок історичних та демографічних подій, таких як розширення популяції з популяцій долин басейну річки Парана–Парагвай, як це було запропоновано мітохондріальною ДНК (де Бріто та ін. 2002). Альтернативно, ця диференціація популяцій може бути пов'язана з локальною адаптацією до різних середовищ за наявності генетичного потоку, оскільки хромосомні інверсії можуть бути пов'язані з адаптивними характеристиками у кактофільних Drosophila (Хассон та ін. 1992; Фернандес Іріарті та ін. 2003). Альтернативне пояснення високої нуклеотидної консервації pBuM-2 у D. antonietae базується на еволюційній динаміці послідовностей супутньої ДНК. Молекулярні механізми, пов'язані з гомогенізацією послідовностей, розташованих в тандемі, такі як нерівний кросинговер та генетична конверсія через процес, званий молекулярним драйвом (Довер 1982; Страчан та ін. 1985), можуть діяти на супутній ДНК pBuM у цьому виді. Наші аналізи не виявили ділянок генетичної конверсії в послідовностях pBuM-2, ізольованих від D. antonietae, що свідчить про те, що цей молекулярний механізм не впливає на різноманітність послідовностей pBuM-2 у цьому виді. Однак було виявлено мінімум 4 події рекомбінації в зразку (Rm = 4) між інтервалами 130–136, 136–155, 174–376 та 376–378. Ці події рекомбінації можуть бути непрямими свідченнями нерівного кросинговеру, що сприяє підтримці первинної структури послідовностей pBuM-2 у різних популяціях D. antonietae. Відповідно до цієї гіпотези, геном D. antonietae містить велику кількість мономерів pBuM-2 (Кун та ін. 2007; Кун та ін. 2008), що підвищує ймовірність нерівного кросинговеру (Сміт1976), і, відповідно, гомогенізації та узгодженої еволюції.
Ці 2 альтернативні гіпотези, що пояснюють високу нуклеотидну схожість, виявлену в мономерах D. antonietae pBuM-2, не є взаємовиключними. У цьому контексті ми пропонуємо, що баланс між генетичним потоком серед популяцій та подіями гомогенізації тандемних масивів супутникової ДНК, спричиненими нерівномірним кросинговером, сприяє збереженню первинної структури pBuM-2 у видах D. antonietae.
Відсутність молекулярної дивергенції в послідовностях супутникової ДНК між популяціями одного й того ж виду, як це було виявлено в даній статті, також спостерігалася в інших видів комах (Bachmann et al. 1994, 1998; Lorite et al. 2002; Feliciello et al. 2005). Наприклад, Bachmann et al. (1994) виявили високі рівні генетичної схожості серед мономерів супутникової ДНК pDoP102 в популяціях Dolichopoda schiavazzii (Orthoptera), які, за даними алозимів, були ізольовані один від одного принаймні протягом 235 000 років. На думку цих авторів, однорідність у pDoP102 навіть в ізольованих популяціях може бути зумовлена біологією супутникової ДНК, оскільки нерівномірний кросинговер і генетична конверсія можуть сприяти підтримці первинної структури послідовностей супутникової ДНК (Bachmann et al. 1994). У іншому дослідженні наявність ділянок генетичної конверсії в мономерах родини супутникової ДНК використовувалася як доказ подій гомогенізації в цій супутниковій ДНК, пояснюючи високу інтраспецифічну консервацію супутникової ДНК, виявлену у фітофагового жука Xanthogaleruca luteola (Lorite et al. 2002).
У кластерному виді D. buzzatii також було виявлено високу інтраспецифічну консервацію послідовності pBuM-2 у D. seriema (Kuhn і Sene 2004) та D. gouveai (Franco, Kuhn та ін. 2006). Більше того, популяції D. buzzatii є однорідними щодо ДНК супутників pBuM-1, ще одного члена родини pBuM (Kuhn та ін. 2003). У D. seriema та D. buzzatii відсутність диференціації популяцій у послідовностях ДНК супутників, як було запропоновано, виникає через минулі та сучасні події генетичного потоку між популяціями, що узгоджується з результатами, отриманими з інших генетичних маркерів, проаналізованих у тих самих популяціях (Kuhn та ін. 2003; Kuhn і Sene 2004). Для
D. gouveai, яка структурована відповідно до моделі «острова» (de Brito et al. 2002; Moraes і Sene 2007), основна гіпотеза для пояснення збереження первинної структури в пBuM-2 супутниковій ДНК полягала в тому, що незалежні механізми гомогенізації, які сприяють повторюваним одиницям, спільним серед популяцій, відбулися після ізоляційної події популяцій D. gouveai (Franco, Kuhn, et al. 2006). Кластер D. buzzatii складається з родинних видів, таксономія яких базується на морфометричних характеристиках адеагуса, що є інтромітентним органом чоловічих статевих органів комах (Vilela 1983; Tidon-Sklorz і Sene 2001; Franco, Prado, et al. 2006). Таким чином, ідентифікація жіночих особин кластера D. buzzatii, отриманих з природного середовища, здійснюється шляхом ідентифікації їхніх чоловічих нащадків, що робить необхідним встановлення ізофемальних ліній, що може бути важким завданням для кактососних Drosophila видів. Більше того, правильна дискримінація видів, заснована лише на кількісних характеристиках, таких як морфологія адеагуса, іноді є складною через наявність морфологічно неоднозначних особин, особливо коли особини збираються в зонах контакту між видами кластера D. buzzatii. У цьому сенсі сильне інтраспецифічне збереження послідовностей супутникової ДНК pBuM, отриманих у кількох дослідженнях (Kuhn et al. 2003; Kuhn і Sene 2004; Franco, Kuhn, et al. 2006; даний документ), навіть у генетично структурованих видах, підкреслює корисність pBuM-2 у вирішенні таксономічних конфліктів серед видів кластера D. buzzatii, визнаного моделі для досліджень еволюційної біології (Manfrin і Sene 2006).
Автори: Фернандо Фарія Франко, Фабіо Мело Сене, Маура Хелена Манфрін
Посилання:
- Арнольд Дж. 1993. Цитонуклеарні дисеквілібрії в гібридних зонах. Annu Rev Ecol Syst. 24:521–554.
- Оберт Дж, Соліньяк М. 1990. Експериментальні докази інтрогресії мітохондріальної ДНК між видами Drosophila. Evolution. 44:1272–1282.
- Бахман Л, Томіук Й, Адис Й, Вохланд К. 1998. Генетична диференціація багатоніжки Pycnotropis epiclysmus, що мешкає в сезонно затоплених і незатоплених амазонських лісах. J Zool Syst Evol Res. 36:65–70.
- Бахман Л, Венанцетті Ф, Сбордоні В. 1994. Характеризація специфічної для виду сім'ї супутникової ДНК Dolichopoda schiavazzi (Orthoptera, Rhaphidophoridae) печерних коників. J Mol Evol. 39:274–281.
- Бетран Е, Розас Й, Наварро А, Барбаділла А. 1997. Оцінка кількості та розподілу довжини трактів генетичної конверсії з даних послідовностей ДНК популяцій. Genetics. 146:89–99.
- Чарльзворт Б, Снєговський П, Штефан В. 1tt4. Еволюційна динаміка повторюваної ДНК у еукаріотів. Nature. 371:215–220.
- де Бріто АР, Манфрін МГ, Сене ФМ. 2002. Нестед кладістичний аналіз бразильських популяцій Drosophila serido. Mol Phylogenet Evol. 22: 131–143.
- Дорадо О, Різеберг ЛГ, Аріас Д. 1992. Інтроґресія хлоропластної ДНК у соняшниках південної Каліфорнії. Evolution. 46:566–572.
- Довер Г. 1982. Молекулярний драйв: згуртований режим еволюції видів. Nature. 299:111–117.
- Елдер ДжФ, Тернер БД. 1tt4. Концертована еволюція на рівні популяції: послідовності супутникової ДНК HindIII у рибок-пупфішів. Proc Natl Acad Sci U S A. 91: 994–998.
- Еккофф'є Л, Лаваль Г, Шнайдер С. 2005. Arlequin (версія 3.0): інтегрований програмний пакет для аналізу даних популяційної генетики. Evol Bioinform Online. 1:47–50.
- Еккофф'є Л, Смауз ПЕ, Кваттро ДжМ. 1992. Аналізи молекулярної варіації, що виводяться з метричних відстаней між гаплотипами ДНК: застосування до даних обмеження мітохондріальної ДНК людини. Genetics. 131:479–491.
- Феліціелло І, Пікаріелло О, Чіналі Г. 2005. Перша характеристика загальної варіабельності повторюваних одиниць у виду виявляє несподівані особливості супутникової ДНК. Gene. 349:153–164.
- Фернандес Іріарт ПЖ, Норрі ФМ, Хассон ЕР. 2003. Хромосомні інверсії впливають на розмір і форму тіла в різних ресурсах розмноження у Drosophila buzzatii. Heredity. 91:51–59.
- Франко ФФ, Кун ГЦС, Сене ФМ, Манфрін МГ. 2006. Збереження послідовностей супутникової ДНК pBuM–2 серед географічно ізольованих популяцій Drosophila gouveai з Бразилії. Genetica. 128:287–295.
- Франко ФФ, Прадо ПРР, Сене ФМ, Коста ЛФ, Манфрін МГ. 200G. Морфологія адеагуса як дискримінантний маркер у двох тісно пов'язаних кактофільних видах Drosophila (Diptera; Drosophilidae) у Південній Америці. An Acad Bras Cienc. 78:203–212.
- Франко ФФ, Сене ФМ, Манфрін МГ. 2008. Молекулярна характеристика сім'ї супутникової ДНК SSS13t у споріднених видах кластера Drosophila buzzatii. Genet Mol Biol. 31:155–159.
- Холл ТА. 1999. BioEdit: зручний редактор вирівнювання біологічних послідовностей та програма аналізу для Windows t5/t8/NT. Nucleic Acids Symp Ser. 41:95–98.
- Хассон Е, Фанара ДжД, Родрігес С, Вілларді ДжК, Рейг ОА, Фонтдевіла А. 1992. Еволюційна історія Drosophila buzzatii. XXIV. Інверсії другого хромосоми мають різний середній ефект на довжину грудей. Heredity. 68:557–563.
- Гудсон РР, Каплан НЛ. 1985. Статистичні властивості кількості рекомбінаційних подій в історії вибірки ДНК. Genetics. 111:147–164.
- Кун ГЦС, Болльгонн С, Сперліх Д, Бахман Л. 1999. Характеризація специфічної для виду супутникової ДНК Drosophila buzzatii. J Zool Syst Evol Res. 37:109–112.
- Кун ГЦС, Франко ФФ, Манфрін МГ, Морейра-Фільйо О, Сене ФМ. 2007. Низькі темпи гомогенізації сім'ї супутникової ДНК DBC-150, обмеженої однією парою мікрохромосом у видів з кластера Drosophila buzzatii. Chromosome Res. 15:457–469.
- Кун ГЦС, Франко ФФ, Сілва ВА молодший, Мартінес-Росі НМ, Сене ФМ. 2003. Про варіабельність супутникової ДНК pBuM18t серед південноамериканських популяцій Drosophila buzzatii. Hereditas. 139:161–166.
- Кун ГЦС, Сене ФМ. 2004. Характеризація та міжпопуляційна варіабельність складної супутникової ДНК HpaI Drosophila seriema (repleta група). Genetica. 121:241–249.
- Кун ГЦС, Сене ФМ. 2005. Еволюційний обіг двох підсімей супутникової ДНК pBuM у кластері видів Drosophila buzzatii (repleta група): від alpha до alpha/beta масивів. Gene. 349:77–85.
- Кун ГЦС, Сене ФМ, Морейра-Фільйо О, Шварцачер Т, Хеслоп-Гаррісон ДжС. 2008. Аналіз послідовностей, хромосомний розподіл і організація на великій відстані показують, що швидкий обіг нових і старих повторів супутникової ДНК pBuM призводить до різних патернів варіації у семи видах кластера Drosophila buzzatii. Chromosome Res. 16:307–324.
- Кумар С, Тамура К, Ней М. 2004. MEGA3: інтегрований програмний засіб для аналізу молекулярної еволюційної генетики та вирівнювання послідовностей. Brief Bioinform. 5:150–163.
- Лоріте П, Каррільо ХА, Гарнерія І, Петієр Е, Паломеке Т. 2002. Супутникова ДНК у жуці листя в'яза, Xanthogaleruca luteola (Coleoptera, Chrysomelidae): характеристика, міжпопуляційний аналіз та локалізація хромосом. Cytogenet Genome Res. 98:302–307.
- Манфрін МГ, Сене ФМ. 200G. Кактофільні Drosophila у Південній Америці: модель для еволюційних досліджень. Genetica. 126:57–75.
- Матейуш РП, Сене ФМ. 2003. Тимчасова та просторово-алозимна варіація у південноамериканській кактофільній Drosophila antonietae (Diptera; Drosophilidae). Biochem Genet. 41:219–233.
- Матейуш РП, Сене ФМ. 2007. Популяційне генетичне дослідження алозимної варіації в природних популяціях Drosophila antonietae (Insecta, Diptera). J Zool Syst Evol Res. 45:136–143.
- Монтейро СГ, Сене ФМ. 1tt5. Морфометричне дослідження популяцій Drosophila serido з Центрального та Південного Бразилії. Rev Bras Genet (Suppl). 18:283.
- Мораес ЕМ, Сене ФМ. 2007. Мікросателітна та морфометрична варіація у Drosophila gouveai: відносна важливість історичних та сучасних факторів у формуванні генетичної структури популяції. J Zoolog Syst Evol Res. 45:336–344.
- Паломеке Т, Лоріте П. 2008. Супутникова ДНК у комах: огляд. Heredity. 100:564–573.
- Перейра МАQR, Вілела КР, Сене ФМ. 1983. Нотатки про місця розмноження та живлення деяких видів групи repleta роду Drosophila (Diptera, Drosophilidae). Cienc Cult. 35:1313–1319.
- Пікаріелло О, Феліціелло І, Беллінеро Р, Чіналі Г. 2002. Супутникова ДНК S1 як таксономічний маркер у коричневих жабах: молекулярні докази того, що Rana graeca graeca та Rana graeca italica є різними видами. Genome. 45: 63–70.
- Руїз А, Кассіан АМ, Кун ГЦС, Алвес МАР, Сене ФМ. 2000. Загадка видоутворення Drosophila serido: складання нових частин разом. Genetica. 108:217–227.
- Сайто Н, Ней М. 1987. Метод сусіднього приєднання: новий метод для реконструкції філогенетичних дерев. Mol Biol Evol. 4:406–425.
- Сембрук Дж, Фрітш ЕФ, Маніатіс Т. 1989. Молекулярне клонування: лабораторний посібник. 2-е вид. Cold Spring Harbor (NY): Cold Spring Harbor Laboratory Press.
- Сміт ГП. 1976. Еволюція повторюваних ДНК послідовностей шляхом нерівного кросоверу. Science. 191:528–535.
- Страчан Т, Уебб Д, Довер ГА. 1985. Перехідні стадії молекулярного драйву в багатокопійних ДНК-сім'ях у Drosophila. EMBO J. 4:1701–1708.
- Томпсон ДжД, Хіггінс ДГ, Гібсон ТД. 1994. CLUSTAL W: покращення чутливості прогресивного множинного вирівнювання послідовностей через вагу послідовностей, специфічні для позицій штрафи за пропуски та вибір матриці ваги. Nucleic Acids Res. 22:4673–4680.
- Тідон-Склорз Р, Сене ФМ. 2001. Два нові види з набору споріднених Drosophila serido (Diptera, Drosophilidae). Iheringia. 90:141–146.
- Угаркович Д, Плохл М. 2002. Варіація в профілях супутникової ДНК — причини та наслідки. EMBO J. 21:5955–5959.
- Вілела КР. 1983. Ревізія групи видів Drosophila repleta (Diptera; Drosophilidae). Rev Bras Entomol. 27:1–114.
- Ватабе Х, Бахман Л, Харінг Е, Сперліх Д. 1997. Таксономічні та молекулярні дослідження Drosophila sinobscura та D. hubeiensis, двох споріднених видів групи D. obscura. J Zool Syst Evol Res. 35:81–94.
- У WL, Ван ДжП, Цзенг МЦ, Чіанг ТЙ. 1999. Клонування та генетична варіабельність повторюваної ДНК HindIII у Acrossocheilus paradoxos (Cyprinidae). Genome. 42:780–788.
