Варіабельність ДНК супутника pBuM189 серед популяцій Drosophila buzzatii в Південній Америці
Машинний переклад
Оригінальна стаття написана мовою EN (посилання для прочитання) .
Супутникова ДНК pBuM189 була проаналізована в популяціях Drosophila buzzatii, які охоплюють більшість ареалу виду в Південній Америці. Ця сатДНК складається з мономерів, багатих на A+T, довжиною 189 пар основ, і попередні дослідження показали швидкий темп еволюційних змін цього компонента геному D. buzzatii. Було вивчено 63 одиниці повторення pBuM189 з 14 популяцій D. buzzatii (9 з Бразилії та 5 з Аргентини). Середня нуклеотидна варіабельність серед 63 повторів становить 4,2%. Принаймні один повтор (Juan/4) здається частиною іншої підсім'ї сатДНК pBuM189. Нуклеотидне вирівнювання всіх 63 повторів не виявило специфічних нуклеотидних замін або інделів, які могли б розрізнити кожну популяцію або групи географічно близьких популяцій. Така відсутність міжпопуляційної диференціації сатДНК узгоджується з попередніми даними mtДНК, які вказують на високий генетичний потік і дуже невелику диференціацію популяцій протягом більшої частини ареалу D. buzzatii в Південній Америці. Генетичний потік міг бути можливим під час льодовикових подій у плейстоцені, таких як та, що відбулася між 13 000 і 18 000 років тому, коли D. buzzatii ймовірно мав більш безперервний ареал, ніж те, що спостерігається сьогодні.
Кактусова муха Drosophila buzzatii (buzzatii кластер, repleta група) має широке поширення в Південній Америці, зустрічаючись на більшості територій Аргентини, Болівії, Парагваю та Бразилії. Вид використовує гнилі тканини кількох кактусів опунції (Opuntia spp.) та колоноподібних кактусів для живлення та розмноження (PEREIRA et al. 1983). Протягом останніх двохсот років кактуси Opuntia були перенесені людиною до кількох регіонів світу, включаючи країни Середземноморського басейну, Африки та Австралії. Відповідно, D. buzzatii мігрувала разом зі своїми господарями. Ця ситуація зробила D. buzzatii відповідним організмом для вирішення питань, пов'язаних з генетичними змінами під час процесу колонізації (FONTDEVILA et al. 1982; HALLIBURTON і BARKER 1993; ROSSI et al. 1996; FRYDENBERG et al. 2002).
Згідно з даними про хромосомні інверсії, аргентинський Чако є найбільш ймовірним центром його радіації (WASSERMAN 1962; CARSON і WASSERMAN 1965). D. buzzatii також дуже поширена в Чако, асоційована з кількома різними видами кактусів (Vilela et al. 1980). Загалом, бразильські популяції розподілені нерівномірно по невеликих ділянках ксерофітної рослинності, де їх було знайдено в дуже низьких частотах (VILELA et al. 1983; TIDON-SKLORZ et al. 1994; TIDON-SKLORZ і SENE 1995). Єдина виняток - південний Бразилія, де були знайдені відносно великі популяції (VILELA et al. 1983; RUIZ et al. 2000).
Було висунуто припущення, що D. buzzatii колонізував Бразилію через зміни в розподілі посушливої рослинності під час льодовикових подій, таких як ті, що відбулися в пізньому плейстоцені (BARKER et al. 1985; FIGUEIREDO and SENE 1992).
BAIMAI et al. (1983) проаналізували метафазні хромосоми кількох популяцій D. buzzatii з Аргентини та Бразилії. Усі відібрані популяції демонструють однакову метафазну пластинку. BARKER et al. (1985) не виявили значних алозимних відмінностей серед південноамериканських популяцій. Лише невелика місцева диференціація, здається, відбулася в бразильських популяціях з Баїї (північний схід) та Траманда́1 (південь). У іншому дослідженні FIGUEIREDO and SENE (1992) повідомили, що з 16 хромосомних інверсій, виявлених в Аргентині, лише дві були присутні в бразильських популяціях, і жодна не була знайдена на північному сході Бразилії. ROSSI et al. (1996) та DE BRITO et al. (2002) виявили високі рівні однорідності mtDNA серед південноамериканських D. buzzatii популяцій, і кілька тестів вказали на високий генетичний потік через більшість географічного розподілу D. buzzatii . DE BRITO et al. (2002) також підрахували, що D. buzzatii присутній у Бразилії щонайменше 100 тисяч років, що свідчить про до-голоценове розширення популяцій D. buzzatii з Аргентини в напрямку Бразилії.
Щоб отримати більше інформації про генетичну варіабельність D. buzzatii в Південній Америці, ми шукали ядерну ДНК. Супутня ДНК (satDNA) є класом високо репетитивної некодуючої ДНК, яка розташована в тандемних великих однорідних масивах у геномі майже всіх еукаріотичних організмів. Зазвичай швидка швидкість еволюційних змін послідовностей satDNA підтримується рядом родин satDNA, які виявилися специфічними для видів (BACH-MANN et al. 1989; KING and CUMMINGS 1997).
Попередні дослідження показали, що послідовності satDNA можуть бути корисними для розрізнення консpecific популяцій (ELDER and TURNER 1994; WU et al. 1999).
KUHN et al. (1999) описали супутню ДНК pBuM189 D. buzzatii. Вона складається з одиниць повторення, які трохи багаті на АТ і мають довжину 189 п.н. П'ять описаних послідовностей pBuM189 були отримані з геномної ДНК лише однієї популяції D. buzzatii. Гібридизаційні та ПЛР експерименти не змогли виявити послідовності pBuM189 у геномі близькоспоріднених видів, таких як D. serido, D. borborema або D. koepferae. Тому доступні дані вказують на швидку швидкість еволюційних змін цього компонента ядерної ДНК D. buzzatii.
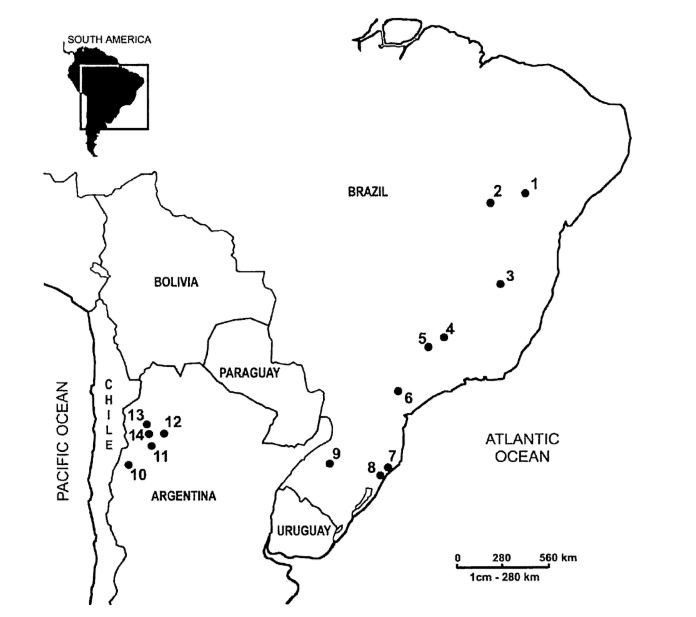
У даному дослідженні ми проаналізували 63 послідовності pBuM189 з штамів, що представляють 14 D. buzzatii популяцій (9 бразильських та 5 аргентинських). Популяції охоплюють більшість географічного розподілу цього виду в Південній Америці.
Матеріали та методи
Зразки
Географічне розташування всіх D. buzzatii популяцій, що були відібрані, показано на рис. 1. Штами, що використовувалися, з їх відповідними місцями: D69R5 (1); J79H41 (2); D54F5 (3); H86G8 (4); D42F2 (5); H99X6 (6); H42F1 (7); J25A20 (8); J28E15 (9); ArgE3 (10); Cat (11), Hondo (12); Salta (13) та Ticucho (14).
Молекулярні техніки
Послідовності pBuM189 були спочатку виділені з геному D. buzzatii після дигестації ДНК ферментом MspI (KUHN et al. 1999). У даній роботі був використаний фермент SspI (який також присутній лише один раз на одиницю повторення pBuM189) для полегшення процедури клонування. Фрагменти satDNA pBuM189 були ідентифіковані за їх появою у вигляді виразних смуг приблизно 190 п.н. на фоні розмиття геномної ДНК. ДНК з смуги ДНК 190 п.н. була елюйована з гелю шляхом нічної інкубації в 500mM NaAc; 1mM EDTA. Відновлені фрагменти були ліговані в вектор плазміди pUC18 і трансформовані в компетентні клітини Escherichia coli DH5-a. Рекомбінантні клони були відібрані у вигляді білих колоній на ампіцилінових пластинах, що містять X-gal (5-бромо-4-хлоро-3-індоліл-β-D-галактозид) та IPTG (ізопропіл-β-D-тіогалактопіранозид). Реакція шаблонної ДНК для секвенування була підготовлена відповідно до інструкції набору BigDye Terminator Cycle Sequencing Ready Reaction Kit (Perkin-Elmer). Автоматичне секвенування ДНК було виконано на секвенаторі ABI Prism™ 377 (Perkin-Elmer).
Для оцінки нуклеотидної варіабельності, генетичних відстаней та побудови NJ дендрограми використовувалася програма MEGA 2.1 (KUMAR et al. 2001).
Результати та обговорення
Як вже очікувалося з попередніх даних послідовності pBuM189, дигестація геномної ДНК 13 D. buzzatii штамів за допомогою SspI дала сильно забарвлену ДНК смугу приблизно 190 п.н., що відповідає мономерам pBuM189, за якою слідували смуги зменшеної інтенсивності сигналів 380 п.н. (димерів), 570 п.н. (тримерів) та інших фрагментів. Смуга ДНК 190 п.н. з кожної дигестації була незалежно елюйована з гелю та клонована. Усього було секвеновано 58 клонів, 39 з 8 бразильських популяцій (місцевості 1 – 4, 6 – 9) та 19 з 5 аргентинських популяцій (місцевості 10 – 14). Нуклеотидне вирівнювання цих 58 клонів з 5 повторів pBuM189 (sert/1 – 5 у даній роботі) з місцевості (5), описаної раніше KUHN et al. (1999), підтвердило, що всі вони представляють мономери satDNA pBuM189 (Рис. 2).

63 повтори pBuM189 є дещо багатими на A+T (71 %) і середня варіабельність послідовності становить 4,2 %. Основним джерелом варіацій серед 63 повторів pBuM189 є однонуклеотидні заміни, які складають 69 % специфічних для повтору, а решта є спільними для двох або більше повторів. Було виявлено лише 11 інделів.
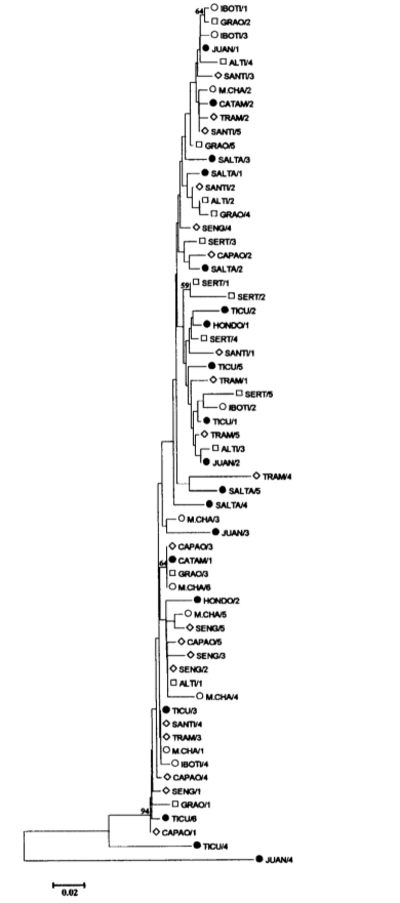
Один повтор pBuM189, здається, є винятковим. Клон Juan/4 з місцевості (10) продемонстрував несподівано високі рівні розбіжності послідовностей у всіх парних порівняннях (в середньому 22,1 % і завжди перевищуючи 19 %). Більше того, тоді як типові послідовності pBuM189 в середньому мають 1,7 % специфічних для повтору однонуклеотидних замін, у повторі Juan/4 було виявлено 30 (наприклад, T на позиціях 11 і 42). Така висока ступінь нуклеотидної розбіжності свідчить про те, що повтор Juan/4 може належати до іншої підродини pBuM189. З теоретичної точки зору, походження підродин satDNA залежить від таких змінних, як кількість копій, геномне розподілення родини satDNA та відносні швидкості нерекіпрокних обмінів всередині та між не гомологічними хромосомами (DOVER 1986).
Якщо повтор Juan/4 виключити з вибірки, середня нуклеотидна варіабельність залишкових 62 послідовностей pBuM189 зменшується до 3.7 %. Це значення майже ідентичне 3.8 %, розрахованому KUHN та ін. (1999) на основі п’яти послідовностей pBuM189, ізольованих лише з однієї популяції D. buzzatii (місцевість 5).
Серед інших повторів pBuM189, Ticu/4 з місцевості (14) є найбільш схожим на Juan/4, оскільки має два специфічні нуклеотиди (G на позиції 70 та A на позиції 88) та один індел (позиція 49), які в іншому випадку є ексклюзивними для повтору Juan/4, а також кілька інших замін, спільних для Juan/4 та інших повторів (наприклад, T на позиції 36 або A на позиції 133). Повтор Ticu/4 сам по собі є найбільш дивергентним повтором pBuM189 (в середньому 10.2 %) після Juan/4, представляючи 6 специфічних нуклеотидних замін повтору (позиції 1, 69, 79, 97, 98, 167 та 188). Тому Ticu/4 слід вважати проміжним еволюційним кроком між «типовими» повторами pBuM189 та сильно дивергентним повтором Juan/4. Цікаво, що обидва рідкі повтори Juan/4 та Ticu/4 походять з Аргентини, де, ймовірно, виник вид (WASSERMAN 1962; CARSON та WASSERMAN 1965).
Згідно з теоретичними та емпіричними даними, диференціація послідовностей satDNA повинна виникати відповідно до концепції консолідованої еволюції (DOVER 1986; ELDER і TURNER 1994). Іншими словами, масиви satDNA з різних популяцій або видів можуть бути гомогенізовані для різних мутацій під впливом молекулярних механізмів, таких як нерівномірний кросинговер і конверсія генів, пов'язані з генетичною ізоляцією та генетичним дрейфом.
Нуклеотидне вирівнювання 63 повторів pBuM189 з 14 D. buzzatii популяцій (в середньому 4.5 % повторів на популяцію) не виявило специфічних нуклеотидних замін або інделів, які могли б відрізнити конкретну популяцію або групи географічно близьких популяцій (Рис. 2). У деяких випадках більше ніж два клони мали ідентичну нуклеотидну послідовність, як у випадку з клонами Capao/3, Grao/3, M.Cha/6 та Catam/1 або M.Cha/1 та Santi/4, але вони походять з досить віддалених географічних локалітетів. З іншого боку, жодних ідентичних повторів не було знайдено в одній популяції.
Генетичні відстані між 63 нуклеотидними послідовностями pBuM189 були розраховані відповідно до методу двох параметрів Кімури (KIMURA 1980), і був побудований дендограм «сусіднього приєднання» (SAITOU і NEI 1987) (Рис. 3). Послідовність Juan/4 була розташована на чітко окремій гілці від інших повторів pBuM189, що підтверджує припущення, що вона належить до іншої підсім'ї satDNA pBuM189. Повтор Ticu/4 був розташований на гілці між Juan/4 та залишковими 61 повтором pBuM189. Серед решти 61 повтору pBuM189 не було спостережено кластерів, що містять послідовності pBuM189 з однієї популяції або груп географічно близьких популяцій.
В цілому, дані вказують на високі рівні схожості послідовностей pBuM189 серед популяцій D. buzzatii, які розташовані, в деяких випадках, на відстані більше 3000 км одна від одної. Така відсутність міжпопуляційної диференціації satDNA узгоджується з даними mtDNA, які вказують на високий генетичний потік і дуже незначну популяційну підструктуру протягом більшості розподілу D. buzzatii в Південній Америці (ROSSI et al. 1996; DE BRITO et al. 2002). Відповідно, повинно бути адаптивне пояснення втрати поліморфізму хромосомних інверсій у бразильських популяціях (в порівнянні з аргентинськими популяціями), про що повідомили FIGUEIREDO і SENE (1992).
Наразі, Чако (північна Аргентина та східна Болівія) та Катаїнга (північний схід Бразилії) є єдиними двома великими районами посушливої рослинності в Південній Америці, які мають високу щільність і різноманітність видів кактусів. В інших районах кактусові менш поширені і розподілені нерівномірно. Те ж саме стосується популяцій D. buzzatii, які залежать від кактусів для харчування та розмноження. Загальновизнано, що глобальний клімат зазнавав повторюваних циклів холодної-сухої та теплої-вологій погоди під час плейстоцену, коли відбулося принаймні чотири основні льодовикові події та багато менших. Наприклад, під час останньої льодовикової події (від 18 000 до 13 000 років тому) клімат у Південній Америці був сухим, і Чако був з'єднаний з Катаїнгою через ксерофітні формації в центральній Бразилії та на атлантичному узбережжі (AB'SABER 1977). Відповідно, D. buzzatii ймовірно мав набагато більш безперервний розподіл, ніж те, що спостерігається сьогодні. Така ситуація могла дозволити генетичний потік серед південноамериканських D. buzzatii популяцій у великій мірі.
На сьогоднішній день більшість супутникових ДНК, описаних у літературі, були ізольовані від осіб з одного штаму або місцевості. Наявні дані є одним з небагатьох прикладів, які надають інформацію про варіабельність послідовностей satDNA серед природних популяцій поширеного виду.
Автори: Густаво С. С. Кун, Фернандо Ф. Франко, Вілсон А. Сілва-молодший, Нільсе М. Мартінес-Россі та Фабіо М. Сенe
Посилання:
- Аб'Сабер, Н. А. 1977. Простори, зайняті розширенням сухих кліматів Південної Америки, під час періодів плейстоцену. – Палеоклімат 3: 1 – 19.
- Бахман, Л., Рааб, М. та Сперліх, Д. 1989. Супутникова ДНК і видоутворення: специфічна для виду супутникова ДНК Drosophila guanche. – Z. Zool. Syst. Evol. Forsch. 27: 84 – 93.
- Баймай, В., Сенe, Ф. М. та Перейра, М. А. Q. Р. 1983. Гетерохроматин і каріотипічна диференціація деяких неотропічних видів кактусів роду Drosophila repleta. – Генетика 67: 81 – 92.
- Баркер, Дж. С. Ф., Сенe, Ф. М., Іст, П. П. та ін. 1985. Алозимна та хромосомна поліморфізм Drosophila buzzatii в Бразилії та Аргентині. – Генетика 67: 161 – 170.
- Карсон, Х. Л. та Вассерман, М. 1965. Поширений хромосомний поліморфізм у поширеному виді, Drosophila buzzatii. – Am. Nat. 99: 111 – 115.
- Де Бріто, Р. А., Манфрін, М. Х. та Сенe, Ф. М. 2002. Філогеографія мітохондріальної ДНК бразильських популяцій Drosophila buzzatii. – Genet. Mol. Biol. 2: 161 – 171.
- Довер, Г. А. 1986. Молекулярний драйв у багатоженевих родинах: як виникають, поширюються та асимілюються біологічні новинки. – Trends Genet. 2: 159 – 165.
- Елдер, Ф. Дж. та Тернер, Б. Дж. 1994. Концертована еволюція на рівні популяцій: супутникові ДНК послідовності HindIII пупфішів. – Proc. Natl Acad. Sci. 91: 994 – 998.
- Фігейредо, В. Л. С. та Сенe, Ф. М. 1992. Варіабельність хромосом у бразильських популяціях Drosophila buzzatii (Diptera, Drosophilidae). – Revta. Bras. Biol. 52: 555 – 561.
- Фонтдевіла, А., Руїз, А., Окана, Дж. та ін. 1982. Еволюційна історія Drosophila buzzatii. II. Наскільки змінився хромосомний поліморфізм під час колонізації? – Еволюція 36: 843 – 851.
- Фріденберг, Дж., Пертольді, К., Дальгард, Дж. та ін. 2002. Генетична варіація в оригінальних та колонізуючих популяціях Drosophila buzzatii, проаналізована за допомогою мікросателітних локусів, ізольованих новим методом ПЛР. – Mol. Ecol. 11: 181 – 190.
- Холлібертон, Р. та Баркер, Дж. С. Ф. 1993. Відсутність варіації мітохондріальної ДНК у австралійських Drosophila buzzatii. – Mol. Biol. Evol. 10: 484 – 487.
- Кімура, М. 1980. Єдиний метод оцінки еволюційної швидкості заміни основи через порівняльні дослідження нуклеотидних послідовностей. – J. Mol. Evol. 16: 111 – 120.
- Кінг, Л. М. та Каммінгс, М. П. 1997. Варіація повторних послідовностей супутникової ДНК є низькою у трьох видах жуків, що ховаються, роду Nicrophorus (Coleoptera: Silphidae). – Mol. Biol. Evol. 14: 1088 – 1095.
- Кун, Г. С. С., Болльгонн, С., Сперліх, Д. та ін. 1999. Характеризація специфічної для виду супутникової ДНК Drosophila buzzatii. – J. Zool. Syst. Evol. Res. 37: 109 – 112.
- Кумар, С., Тамура, К., Якобсен, І. Б. та ін. 2001. MEGA2: програмне забезпечення для молекулярного еволюційного генетичного аналізу. – Університет штату Аризона.
- Перейра, М. А. Q. Р., Вілела, К. Р. та Сенe, Ф. М. 1983. Примітки про місця розмноження та живлення деяких видів групи repleta роду Drosophila (Diptera, Drosophilidae). – Наука і культура 35: 1313 – 1319.
- Россі, М. С., Барріо, Е., Латорре, А. та ін. 1996. Еволюційна історія Drosophila buzzatii. XXX. Поліморфізм мітохондріальної ДНК в оригінальних та колонізуючих популяціях. – Mol. Biol. Evol. 13: 314 – 323.
- Руїз, А., Кансіан, А. М., Кун, Г. С. С. та ін. 2000. Головоломка видоутворення Drosophila serido: збирання нових частин. – Генетика 108: 217 – 227.
- Сайто, Н. та Ней, М. 1987. Метод сусіднього приєднання: новий метод для реконструкції філогенетичних дерев. – Mol. Biol. Evol. 4: 406 – 425.
- Тідон-Склорц, Р. та Сенe, Ф. М. 1995. Фауна Drosophila (Diptera, Drosophilidae) у північній частині «Cadeia do Espinhac¸o'', штатів Мінас-Жерайс і Баїя, Бразилія: біогеографічні та екологічні аспекти. – Iheringia Ser. Zool. 78: 85 – 94.
- Тідон-Склорц, Р., Вілела, К. Р., Сенe, Ф. М. та ін. 1994. Рід Drosophila (Diptera, Drosophilidae) в Серра до Чіпо, штат Мінас-Жерайс, Бразилія. – Revta. Bras. Ent. 38: 627 – 637.
- Вілела, К. Р., Сенe, Ф. М. та Перейра, М. А. Q. Р. 1980. Про фауну Drosophila Чако та східних схилів Анд в Аргентині. – Revta. Bras. Biol. 40: 837 – 841. Вілела, К. Р., Перейра, М. А. Q. Р. та Сенe, Ф. М. 1983. Попередні дані про географічний розподіл видів Drosophila в межах морфокліматичних доменів Бразилії. II. Група repleta. – Ciên. Cult. São Paulo 35: 66 – 70.
- Вассерман, М. 1962. Цитологічні дослідження групи repleta роду Drosophila. V. Підгрупа mulleri. – Univ. Texas Publ. 6205: 85 – 118.
- У, В. Л., Ван, Дж. П., Цзенг, М. С. та ін. 1999. Клонування та генетична варіабельність повторюваної ДНК HindIII у Acrossocheilus paradoxus (Cyprinidae). – Genome 42: 780 – 788.